Saúde
Mapa novo e mais detalhado da resistência antimicrobiana
Durante a pandemia de COVID-19, o mundo se conscientizou do valor de usar análises de esgoto para monitorar o desenvolvimento de doenças em uma área. No entanto, no DTU National Food Institute, um grupo de pesquisadores usa o monitoramento...
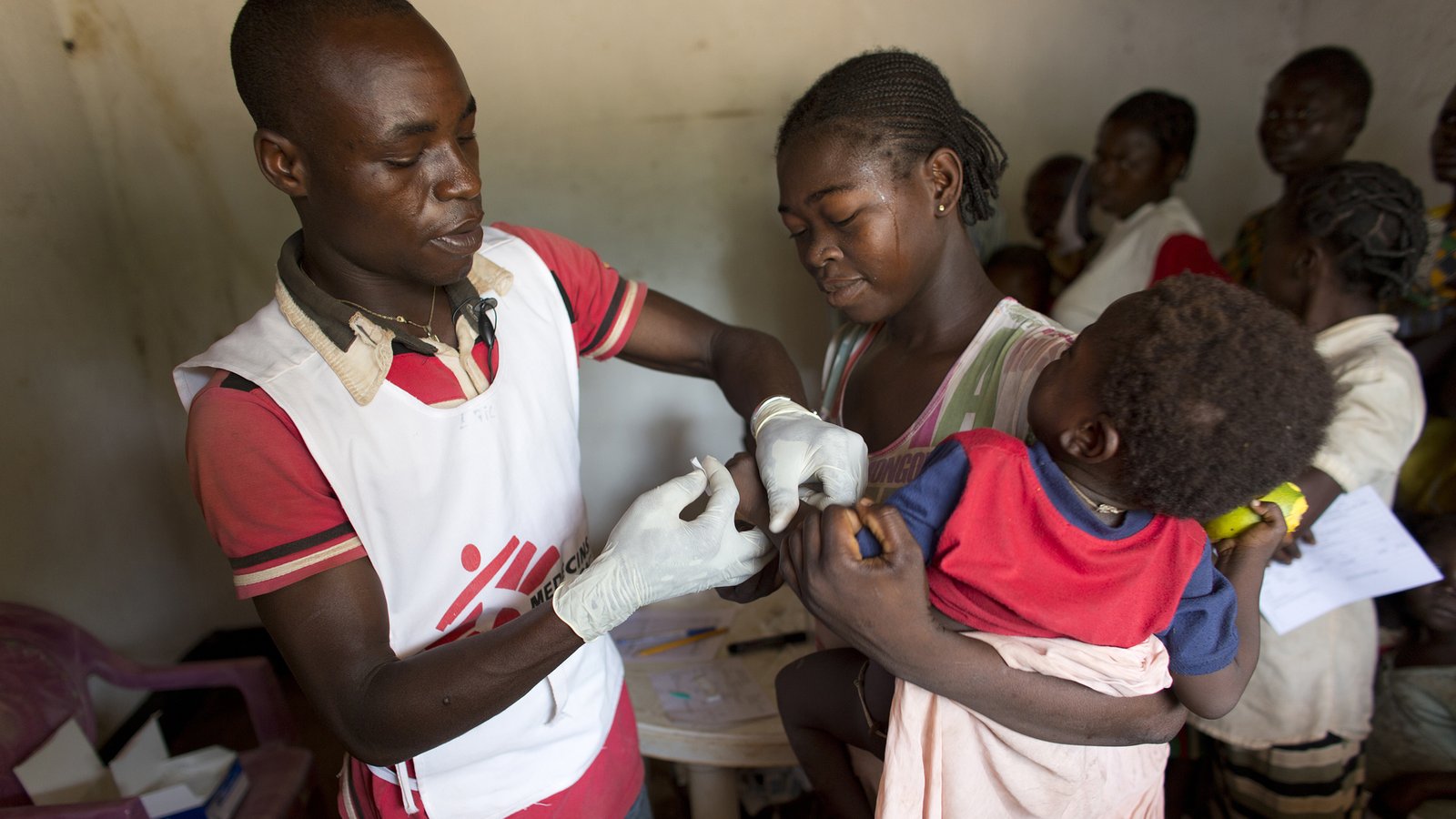
Coleta de amostra de esgoto em Tamale, Gana - um dos 101 países que participaram de uma vigilância global de doenças infecciosas e resistência antimicrobiana via esgoto. Crédito: Coragem Kosi Setsoafia Saba
Durante a pandemia de COVID-19, o mundo se conscientizou do valor de usar análises de esgoto para monitorar o desenvolvimento de doenças em uma área. No entanto, no DTU National Food Institute, um grupo de pesquisadores usa o monitoramento de esgoto de todo o mundo desde 2016 como uma ferramenta eficaz e barata para monitorar doenças infecciosas e resistência antimicrobiana.
Ao analisar amostras de esgoto recebidas pela DTU de 243 cidades em 101 países entre 2016 e 2019, os pesquisadores já mapearam onde no mundo a ocorrência de genes de resistência é maior, como os genes estão localizados e em quais tipos de bactérias eles são encontrados .
Os resultados do novo estudo metagenômico – que acaba de ser publicado na Nature Communications – surpreenderam os pesquisadores. Na verdade, o estudo mostra que os genes apareceram em muitos contextos genéticos e tipos bacterianos diferentes, indicando uma transmissão maior do que os pesquisadores esperavam.
"Encontramos genes de resistência semelhantes em tipos bacterianos altamente diferentes. Achamos preocupante quando os genes podem passar de um grupo muito amplo de bactérias para um grupo completamente diferente com o qual não há semelhança. É raro que essas transmissões de genes ocorram ao longo distâncias tão longas. É um pouco como espécies animais muito diferentes produzindo filhotes", explica o professor assistente Patrick Munk.
Se os genes estiverem em bactérias que geralmente não causam doenças - como as bactérias do ácido lático -, isso é menos preocupante. No entanto, se os genes de resistência se infiltrarem em bactérias importantes para a saúde humana – como a salmonela – a história é completamente diferente.
"Isso torna muito mais provável que a bactéria realmente mate pessoas - por exemplo, em um hospital - porque não há tratamento disponível", diz Patrick Munk.
Como um quebra-cabeça intrincado
O Grupo de Pesquisa para Epidemiologia Genômica do DTU National Food Institute desenvolveu e mantém um dos bancos de dados de resistência mais abrangentes do mundo. Atualmente inclui 3.134 genes de resistência conhecidos.
Os pesquisadores usaram o banco de dados para mapear genes de resistência nas amostras de esgoto no novo estudo.
As amostras contêm um número muito grande de microorganismos de diferentes origens, incluindo fezes humanas. As amostras de esgoto congeladas foram enviadas para a DTU, onde os técnicos do laboratório extraíram todas as bactérias das amostras descongeladas.
As bactérias são então quebradas e seu DNA coletivo é quebrado em pedaços menores , que equipamentos de sequenciamento de DNA de última geração podem ler de uma só vez.
Um supercomputador pode então comparar os bilhões de sequências de DNA registradas com genes conhecidos e construir pedaços maiores dos genomas originais contidos nas amostras.
Esse processo fornece informações sobre várias áreas, como em quais bactérias e vizinhanças genéticas os genes de resistência estão localizados.
O mapa mundial mostra a ocorrência de genes de resistência em diferentes países.
Quanto mais escura a cor usada para um país, maior a frequência de genes de
resistência nas bactérias. Os países cinzas não forneceram amostras de esgoto.
Crédito: Universidade Técnica da Dinamarca
Hotspots para transmissão de genes
Em diferentes lugares da África Subsaariana, os pesquisadores encontraram o mesmo gene de resistência em várias bactérias diferentes.
"Nós interpretamos isso como significando que podemos estar bem próximos de um ponto de transmissão, onde há uma transmissão de genes de uma para outra para uma terceira bactéria. É por isso que estamos vendo o gene em tantos contextos diferentes precisamente ali", disse Patrick Munk explica.
Ele acrescenta que muitas das transmissões surpreendentes parecem ocorrer na África Subsaariana. Estes são também os países com programas menos desenvolvidos de monitoramento da resistência, o que significa que há muito poucos dados sobre a situação da resistência.
“Corremos o risco de ignorar tendências importantes porque não temos dados”, sugere, enfatizando que dados sólidos são exatamente o que é necessário para desenvolver estratégias eficazes de combate à resistência:
"Neste momento, temos um grande conhecimento sobre como a resistência se comporta no Ocidente e - com base nesse conhecimento - planejamos como combater a resistência. Acontece que, se olharmos para alguns novos locais, os genes de resistência podem se comportar de maneira muito diferente - presumivelmente porque eles têm condições de transmissão mais favoráveis. Portanto, a maneira como você combate a resistência também deve ser ajustada e adaptada às condições locais."
Sucessor
O projeto global de esgoto – apoiado pela Fundação Novo Nordisk e pelo projeto de pesquisa VEO – termina em 2023. Os pesquisadores acham que ele provou ser um bom complemento para as iniciativas de monitoramento existentes, que operam principalmente em nível nacional ou regional e medem resistência em bactérias de pessoas doentes.
Esperam, portanto, que apareça um sucessor do projeto, para que o mundo continue se beneficiando do importante conhecimento gerado pelo programa de monitoramento. Isso também se aplica a países que possuem programas sólidos de monitoramento e estratégias de controle em vigor.
"Existem muitas analogias com as mudanças climáticas, onde o que acontece do outro lado do globo não é importante para você. Um dia ou outro, o problema voltará a nos atormentar, como já vimos várias vezes." Patrick Munk enfatiza.
Dados reutilizáveis
Ao contrário dos dados dos métodos de análise convencionais, os dados brutos dos estudos metagenômicos podem ser reutilizados para esclarecer outros problemas. Por exemplo, os pesquisadores do projeto de esgoto usaram seu conjunto de dados para analisar a ocorrência de outros microrganismos patogênicos no esgoto.
Todo o conjunto de dados do monitoramento de esgoto foi disponibilizado gratuitamente para pesquisadores em todo o mundo. Por exemplo, já foi usado para detectar muitos novos vírus globalmente e para mapear a composição étnica de diferentes populações.
À medida que novos genes de resistência são descobertos - mesmo no futuro distante - os pesquisadores poderão reutilizar dados brutos para estabelecer rapidamente onde eles apareceram pela primeira vez e como se espalharam.
No estudo, os pesquisadores analisaram 757 amostras de esgoto de 243 cidades em 101 países. As amostras foram coletadas e enviadas ao campus da DTU em Lyngby entre 2016 e 2019.
A análise genômica de águas residuais é rápida e razoavelmente barata em relação a quantas pessoas você pode cobrir. As análises de águas residuais não requerem aprovação ética, pois o material da amostra não pode ser vinculado a indivíduos.
Mais informações: Patrick Munk et al, Análise genômica de esgoto de 101 países revela cenário global de resistência antimicrobiana, Nature Communications (2022). DOI: 10.1038/s41467-022-34312-7
Informações do jornal: Nature Communications